

近年、目覚ましい発展を続ける生命科学。そのなかでも、蛋白質や核酸などの生体高分子の 構造に基づく機能解析は、最も注目される研究領域である。
大阪大学蛋白質研究所では、生命科学の応用研究を支える蛋白質の基礎研究に取り組んでいる。同施設に設置されている日本蛋白質構造データバンク(PDBj)は、世界に4つある蛋白質データバンクの国際研究機関の一つとして、日々、蛋白質の立体構造データを登録・編集し、 情報の提供を行っている。「蛋白質のデータ科学」が見据える生命科学の未来像について、大阪大学蛋白質研究所・所長の中村春木教授に伺った。

大阪大学の吹田キャンパス、「大阪大学蛋白質研究所」内にある「日本蛋白質構造データバンク(PDBj)」は、米国のRCSB-PDB、BMRB、EUのPDBeとともに世界の4拠点の一つとして、主にアジアの研究者から寄せられた膨大な量の蛋白質の3次元構造データの登録・編集・公開を行っている。中村教授はPDBjのトップとして、米国と欧州のPDBとテレビ電話で連絡をとりあいながら、協調して運営を行っている。
近年、電子顕微鏡の高性能化をはじめとする技術革新により、原子レベルで詳細に蛋白質の構造を見ることが可能となった。人工的に蛋白質を合成、改造する方法も進んでおり、そのような「蛋白質工学」は、創薬や医療、化学産業、化粧品や洗剤など身近な製品の開発でも、先端技術として成長を続けている。
蛋白質工学の基礎研究で、日本をリードしてきたのが大阪大学だ。大阪大学では第二次大戦以前から、理学部と医学部を中心として蛋白質の研究がさかんに行われていた。1958 年には全国の研究者の共同利用研究所として、「大阪大学蛋白質研究所」が設置され、以来、日本の蛋白質研究の中心的役割を担ってきた。
その大阪大学蛋白質研究所に、「日本蛋白質構造データバンク(PDBj:Protein Data Bank Japan)」が創設されたのは、2000年7月のことだ。PDBj は、米国に2カ所、欧州に1カ所 あるデータバンクのメンバーの一員として、おもにアジア地域の構造生物学研究者から蛋白質の立体構造データを受け入れ、登録作業を行い、品質の高い共通の国際的データベースの構築・運営を行っている。
「蛋白質構造データバンク」には、各国の研究者の手によって、原子ひとつひとつの位置がわかるレベルで解明された蛋白質の構造情報が膨大に登録されている。その解析手法は、X線結晶回析、NMR(核磁気共鳴法)、クライオ電子顕微鏡などさまざまだ。これらの構造情報は、研究者・教育者・学生・企業を問わず、誰でも無償で利用することができる。2015年には5億3千万件もの登録情報のダウンロードが行われており、生命科学の研究者にとってPDBの情報は必須の研究資料となっている。
自然界に約10万種類存在すると言われる蛋白質は、生物の細胞のなかでDNA(デオキシリボ核酸)の遺伝情報に従ってアミノ酸が鎖状に結合し、3次元の立体構造に折り畳まれる。その構造は柔らかく、同じ蛋白質でも、パートナーとなる別の蛋白質など相手分子との結合の仕方によって、また時間の経過とともに形が変化する(下図参照)。そして、構造が変化することによって、生物が生きるために必要なそれぞれの機能を発揮する。つまりすべての生命活動の根幹には蛋白質の構造があり、その構造の解明は、すなわち生命の秘密に迫ることに直結するのである。
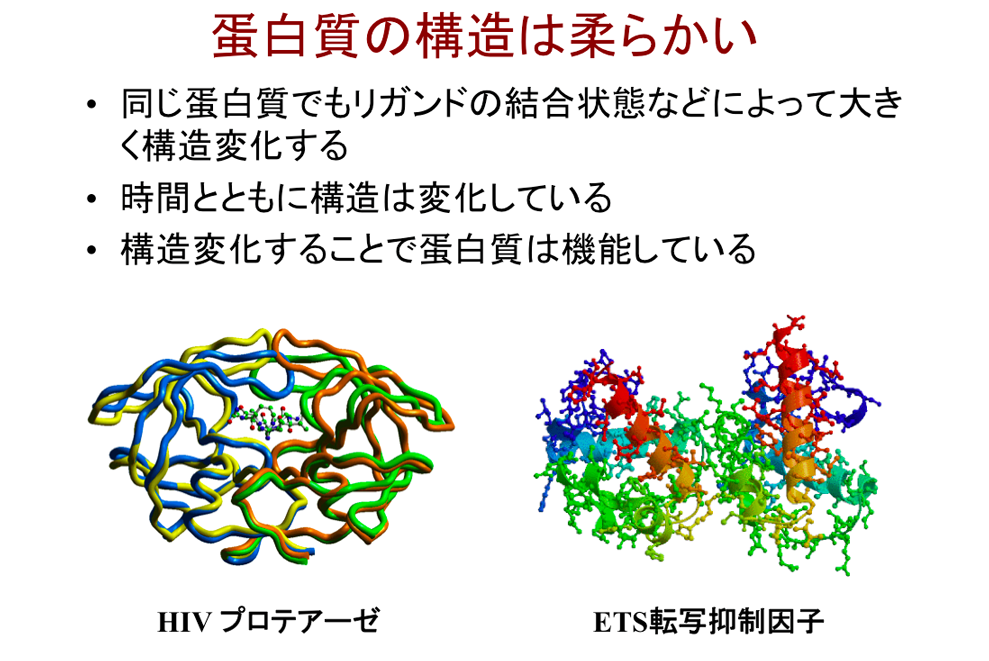
蛋白質は柔らかく、種類ごとにさまざま形を示すだけでなく、時間の経過によっても形が変化する。図の「HIVプロテアーゼ」「ETS転写抑制因子」はそれぞれヒト免疫不全症候群(AIDS)、癌に密接に関わり、構造の特定が新薬の開発へとつながっている。
大阪大学蛋白質研究所の所長であり、PDBjの責任者を務める中村春木氏は、蛋白質構造データバンクの意義について次のように語る。
「こうした蛋白質の構造に関する知識はいわば『人類の宝』です。そこで『各研究者が知見を溜め込まず、広く一般にも公開することにしよう』とデータベースを作る動きがアメリカで1970年代に起こりました。それが、生体高分子の構造に関するすべての情報を集める国際機関『蛋白質構造データバンク(PDB)』創設のきっかけとなりました」
最初はたった7個の登録で始まった蛋白質の構造情報だが、現在では約12万件、毎週数百件のペースでデータの更新が行われている。近年ではこのデータバンクに登録することが論文発表の条件に定められたこともあり、世界中の生命科学の研究者が、このデータバンクを第一資料として登録・利用している。人間の体を構成する蛋白質はおよそ6万種類あると言われているが、そのうち約7割程度の構造がPDBにカバーされるようになっている。
中村教授は蛋白質研究所の所長として施設の運営全体の責任者を務めると同時に、自身も「蛋白質データ科学」の国内第一人者として研究を続ける。教授は、蛋白質研究の最先端では、自然科学のあり方を変える大きな変化が起きていると語る。
「これまで、自然科学の世界には4つの大きなパラダイム・シフトが起きています。1番目は、古代ギリシャで星を観察することで生み出された『天動説』のように、経験にもとづいて自然を記述する考え方。2番目は、ニュートンが発見した万有引力の法則に代表される、数学の微分や積分を応用した理論と実験によって自然界を記述するやり方。3番目が理研の『京』に代表されるスーパーコンピュータによって、複雑な自然現象をシミュレートしCGなどで可視化して理解する方法。そして最後の4番目、現在進行中の新たなパラダイムとして考えられているのが『ビッグデータを活用したデータ科学』です。蛋白質研究も、この『データ科学』のパラダイムに突入しています」
「データ科学」は、「データ主導科学」あるいは「データ駆動科学」などとも呼ばれる。この新しいパラダイムでは、散在している生の「データ(Data)」を「情報(Information)」として整理し、それを「知見(Knowledge)」にまとめあげ、「知見」の蓄積を進めることで、問題解決につながる「智慧(Wisdom)」を構築する。これが、「DIKWピラミッド」と呼ばれる一連のプロセスだ。コンピュータの進歩により、莫大な量の「情報」をAI技術(人工知能)と結びつけることで、初めて可能となった研究手法だ(右上の図参照)。
ただし、蛋白質の研究でデータ科学を導入するためには、三つの注意すべき点があると、中村教授は指摘する。「正しいデータ」のみを扱うこと、「最新の技術」にもとづいた蛋白質の構造情報であること、そして、最新の技術にもとづいた正確なデータが存在しない場合はそれを「生み出す」ことだ。
「ビッグデータ研究では、非常に膨大な量のデータを扱います。そのなかに、精度の低いデータが少々含まれていても、誤差として吸収されるのが通例です。ところが、我々の扱う生物の領域のサイエンスでは、実験数値の桁が一つ間違っていたら、出てくる結果に重大な影響を与えます。だからこそ、扱うデータが厳密に正確であるよう、常にデータの正しさを客観的にチェックする仕組みが必要となるのです」
PDBでは、研究者が先を争うようにしてデータが登録される。だが、そのままでは玉石混交で、研究には適さない。きちんと登録条件を満たしているか、専門の「アノテータ」と呼ばれる人の目でチェックをすると同時に、構造データの裏付けとなる実験データの付与も義務づけられている。後でデータをユーザが安心して使用するためには、この水際の作業が非常に重要となる。
データがいつ、どのような技術で得られたものかもやはり重要だ。蛋白質の構造情報を取得する技術は、日々進歩を続け、精度も高まっている。「最新の技術」がデータの正しさを裏付ける有力な根拠となる。そして、この2つの条件を満たすデータが存在しない場合は、それを研究者自ら「生み出す」姿勢も欠かせない。そのための実験技術も、コンピュータ・シミュレーション手法も日進月歩で新しいものが生まれている。それらを駆使してデータを生み出すことが、生命科学を前に進める原動力になるのだ。
これまでの科学は、何かしらの現象が先にあり、その原因や理路を解き明かすという順番で理論が構築されてきた。データ科学ではその逆に、先に構造や動きのデータを収集し仮説を立てて、あとからそれを実験により検証するというプロセスをとる。

本年2016年の春に蛋白質研究所に導入されたばかりの最新鋭クライオ電子顕微鏡。対象物を液体ヘリウムで超低温に冷やし、さまざまな方向を向いた沢山の粒子を計測して、単粒子回析とよばれる手法を用いてデータを統合することで、物体の形状を原子レベルで微細に見ることができる。
先にも触れたように、人間を構成する蛋白質のうち、約7割の三次元構造が解明され、PDB に登録されている。なかには、人以外のバクテリアや猿、マウスなどと共通する「ホモログ」と呼ばれる蛋白質の構造も含まれるが、いずれにしろかなりの部分が解明されてきたのは確かだ。だが、残りの3割の解明には、大きな課題が残されていると中村教授は語る。
「残る3割の蛋白質の大部分を占めるのが、『天然変性蛋白質』と呼ばれる、特定の形を持たない蛋白質です。天然変性蛋白質は、普段は不定形でふにゃふにゃした形をしていますが、パートナーとなる蛋白質と結合するなどの相互作用が働くと、特定の形をとって機能を果たします。構造のフォールディング(折り畳み)とバインディング(結合)が同時に起こる、こうした蛋白質の柔らかな構造については未だによくわかっていません。しかもこれらは、近年の生物学で非常にホットなトピックとなっているエピジェネティクス (生物のゲノムの情報によらず蛋白質が発現したあとで修飾される変化)と密接に関わっていることが判明し、注目が高まっているのです」

クライオ電子顕微鏡で観察した蛋白質の画像。右の白黒の画像に多数映っている細胞の方向や向きを揃えてデータ処理することにより、左のような原子一つ一つの位置まで確認できる詳細な画像が得られるようになった(画像は、蛋白質研究所の岩崎憲治准教授、宮崎直幸特任助教、中川敦史教授によって解析されたもの)。
天然変性蛋白質はもともとの形が不定形であるため、従来観察手法として使われてきたX線構造回折では、必要な結晶構造をつくるのが難しい。そこで行われるのがスーパーコンピュータを使った「分子動力学計算」と呼ばれるシミュレーションである。この計算方法はもともと、1977年に米理論科学者のマーティン・カープラス教授らのグループがネイチャーに発表した技術で、複雑な蛋白質や生体分子の振る舞いを、物理法則に基づいてコンピュータ上でシミュレーションする方法を切り拓いた。その功績でカープラス教授は2013年のノーベル化学賞を受賞している。
「これにより、例えばエピジェネティクスではよく『リン酸化して活性化する』と言いますが、実際にそのとき蛋白質がどのような形をとっているか、CGで見て構造を理解できるようになりました。最近ではコンピュータで10億ステップにもなる特殊な分子動力計算を行い、非常に大きな蛋白質の動きもシミュレーションをすることが可能となっています。こうした計算によって生み出した構造も、データとして扱えるまでに信頼性が向上したことから、データ科学の3つ目の課題としてデータベース化する動きが始まっています」
こうした手法はすでに創薬にも応用されている。疾病の原因となる蛋白質とその立体構造を同定し、その蛋白質を標的とする新しい医薬品を、シミュレーション計算によって何百万種類におよぶ候補の化合物の中から選び出し、合成・設計するのである。この「インシリコ(in silico)創薬」と呼ばれる手法は1980年代から始まり、現在の創薬の中心技術となりつつある(「in silico」とは「コンピュータ内で」という意味。通常の生物を対象とする実験で言われる「in vivo(生体内で)」や、「in vitro(試験管内で)」という言い方と対照的に用いる)。
「ゲノム情報がわかれば、その情報を元に蛋白質の立体構造を推定し、薬が効くかどうかも予測できます。我々の研究所では、インフルエンザ薬のタミフルが効く原理を、インフルエンザウィルス中の蛋白質の構造から解き明かしました。インフルエンザウィルスの表面には、ノイラミニダーゼという酵素があります。そのアミノ酸配列274番目にあるヒスチジンというアミノ酸がチロシンというアミノ酸に変わると、タミフルが効かなくなるという現象が確認されていました。なぜそれが効かなくなるのかは不明だったのですが、我々がベトナムの研究者とともに行ったシミュレーションの結果、蛋白質の構造が変化することによって、チロシンにタミフルが結合しなくなることが、目に見える形で明らかになったのです」
この他にも中村教授らは、大手製薬メーカーと共同で、副作用が少なくより効果のある鎮痛剤の開発なども進めている。免疫反応の研究においても、抗体が抗原を認識するプロセスの解明に蛋白質データ科学は活用されており、医療分野の研究で欠かせない手法となっている。
中村教授はもともと東京大学の理学部物理学科の出身で、同大学院で生物物理の領域に足を踏み入れた。以後、一貫して蛋白質の構造研究の道を歩み、イギリスへ留学した1984年に、 蛋白質工学という新しい学問分野の潮流と出会った。
世界的な蛋白質研究の盛り上がりの波を受け、日本でも大阪の吹田に通産省の指導のもと 「蛋白工学研究所」(現・理化学研究所・生命システム研究センター)が設置され、中村教授は東京大学からそこに籍を移した。そしてコンピュータを用いて、情報科学や計算科学を応用して蛋白質を分析する「構造バイオインフォマティクス」の国内第一人者として、研究を進めていった。
「私が大学院生のときは、東京大学の計算機センターで、黎明期の蛋白質データベースを使う側の人間でした。その頃は当然インターネットもなく、記録媒体は磁気テープの時代です。 その後の蛋白工学研究所ではデータを生産し解析する側にまわって、現在ではデータベースを運営する立場となりました」
これからの蛋白質研究所の目標として、中村教授は「生命現象をマルチスケールで理解することです」と述べる。
「蛋白質はすべての生命現象の源です。病気のときに薬が効いて、症状が良くなるということの根本も、薬の化学物質が蛋白質と結合して構造が変化し、その結果、蛋白質が果たしていた機能がストップしたり、あるいはより強められたりすることで、生体に変化が起こるわけです。複雑な生命現象は、その蛋白質同士の相互作用のプロセスが分かってはじめて完全な理解に到達できます。細胞や細胞内の蛋白質の動きを、さまざまな観測手法を組み合わせて原子レベルで解明しながら、統合的な生命現象の理解へとつなげていくことが、今まさに我々がやるべきことだと思っています」
そのために蛋白質研究所では、研究所内に新しく『多階層蛋白質統合研究部門』を創設。 最新鋭のクライオ電子顕微鏡をはじめとする様々な手法によるマルチスケールの研究が、今まさに始まったところだ。
物理学のバックボーンから生命科学へと研究の領域を移した中村教授は、「生命現象を、物理の視点で解き明かすことにこの研究の面白みを感じる」と語る。生命現象も原子レベルでは物理法則にもとづいて起きており、だからこそデータ科学やシミュレーションで、実際の蛋白質の動きを再現できる。だがその解明のためには、コンピュータのプログラミングや、 最新の実験手法といった種々のスキルを会得しておく必要がある。中村教授も「研究者は若いときから、さまざまなスキルを意識して身につけておくと良いでしょう」とアドバイスする。
「『これを解き明かしたい』というサイエンスの目標を持ち、興味があることに、長期的に諦めず向き合い続けていれば、やがてそれらのスキルが自分の武器となり、研究の突破口を開いてくれるはずです」
蛋白質の構造解明を通じて生命の秘密に迫る大阪大学蛋白質研究所が、これからどのような成果を生み出してくれるのか、楽しみでならない。


1958 年に全国共同利用研究所として設立。4 研究部門13 研究室と1センター7研究室に加えて2つの寄附研究部門(ニッピおよび日本電子)からなる体制を整え、日本における蛋白質研究の中核「蛋白質研究共同利用・共同研究拠点」として国内外から多くの研究者が集まる。2000年からは蛋白質立体構造データバンク(PDB)の世界4 拠点の一つとして PDBj を運営し、主にアジア地区のデータ登録や世界に対する種々のサービスを行う。
【取材・文:大越裕/撮影:トヨサキジュン】
 bana1.png (300px×80px)
bana1.png (300px×80px) bana1_e.png (300px×80px)
bana1_e.png (300px×80px) NovelPrize2015.png
NovelPrize2015.png